人类基因组中单个或多个核苷酸的突变,是导致遗传性健康问题的主要根源[1]。现有统计表明,单基因突变已引发超过7000种被确认的遗传性疾病[2]。针对这类基因突变引发的疾病,治疗手段已从传统的酶替代和小分子药物,拓展至体内/体外基因治疗及细胞移植等多层次、多模式的精准医学方案。
基因治疗(Gene Therapy)指通过向患者体内递送功能性基因或编辑现有基因,从而纠正致病突变、补充缺失基因或调控异常通路,从根源上干预疾病的治疗策略。该策略既可运用基因补充方法——例如利用病毒或非病毒载体将正常基因导入细胞,也可通过CRISPR/Cas、TALEN、ZFN等基因编辑技术直接修改基因组。基因编辑领域的一项重大突破在于发现可作为"分子剪刀"的核酸酶。这类酶使基因编辑技术能在活细胞内精确定位并修饰特定DNA/RNA序列,为纠正遗传性疾病开辟了新途径。
基因编辑技术的引入推动了分子生物学和生物技术的蓬勃发展,开启了后基因组时代。在过去十年间,多种核酸酶基因编辑技术相继被开发并得到广泛应用。本文着重介绍了几种备受关注的基因编辑技术的作用机制以及应用。
一、巨核酸酶(Meganuclease)
巨核酸酶(Meganuclease),也被称作大范围核酶,属于核酸内切酶,利用蛋白侧链氨基酸的氢键或范德华相互作用直接识别 DNA碱基的化学基团,其对DNA靶位点的识别与切割序列较长,一般为12-45bp [3]。天然存在的巨核酸酶主要以归巢核酸内切酶为典型代表。归巢核酸内切酶通常由表现为可移动遗传元件的内含子或内含肽编码,它们能够识别无内含子或无内含肽的同源等位基因,并在该位置产生双链断裂(DSB)。通过与含有内含子或内含肽的等位基因进行同源重组来修复DSB,会导致内含子或内含肽插入到DSB发生之处。这一过程促进了生物体内遗传元件的横向移动,该过程被称为“归巢” [4]。
巨核酸酶的使用优势与限制:①MegN拥有较长的识别位点,对靶DNA具备高度特异性;②目前,天然大范围核酸酶的天然库仅包含约300种蛋白质,其中大多数仍为假设性的或未经过表征。这意味着它能够识别的靶位点相对较少,从而限制了其适用范围[5][6]。
![]()
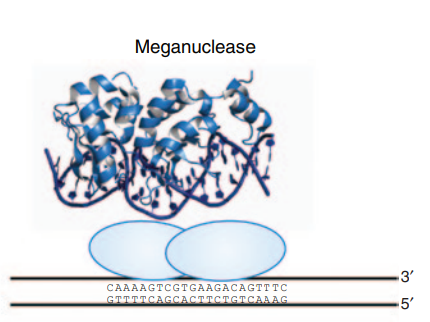
图1:巨核酸酶示意图(Maeder,M.L.,&Gersbach,C.A.,Molecular therapy,2016)
二、锌指核酸酶(ZFNs)
锌指蛋白(Zinc Finger Proteins,ZFPs)是人工合成的工程化杂合异二聚体蛋白,每个结构域可与3 - 6个碱基对的特定序列相结合[7]。锌指(Zinc - Finger)是一类能够结合锌离子(Zn²⁺)的结构域,其折叠方式使蛋白质链在该区域形成类似手指的突起。每根“指”由特定的氨基酸序列(常见的C2H2型为Cys- Cys-His-His)与锌离子配位,从而形成一个稳定的“指状”结构。这些指状结构能够实现对特定DNA序列的识别与结合。正是由于这种“指”形的DNA结合方式,研究者将其称为“锌指”。
锌指核酸酶(Zinc-Finger Nuclease,ZFN)由DNA识别域(锌指结构)和核酸酶切割域(FokI)组成。两个杂合异二聚体锌指蛋白与两个FokI异二聚体核酸酶(ELD/KKR)融合[8],两个ZFPs单体分别结合相邻的DNA序列,形成“左-右”配对后,FokI切割域相互靠近并二聚化,切割DNA产生双链断裂(DSB)。细胞随后通过非同源末端连接(NHEJ)或同源重组(HDR)修复,实现基因敲除、敲入或碱基替换[9]。
锌指核酸酶的锌指蛋白可通过蛋白工程化,使每个锌指识别3个碱基,多个锌指蛋白组合后可实现长序列特异性结合,同时通过开发FokI核酸酶结构域变体,从而增加该系统的结合和切割的特异性,降低其细胞毒性提高临床应用的安全性。
锌指核酸酶的使用限制与优势:①由于锌指核酸酶的设计成本和技术门槛较高,在大规模基因筛选中,它不如 CRISPR 便捷。②然而,随着锌指模块库的不断完善和 FokI 结构的持续优化,凭借高特异性以及对体内递送友好的优势,锌指核酸酶已在基因治疗、动物模型构建和作物育种等多个领域取得了实质性进展,并且在对脱靶要求极高的临床场景中保持着竞争力。
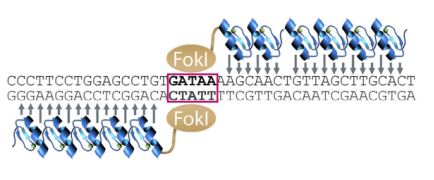
图2:锌指核酸酶示意图(Lessard, Samuel et al., Scientific reports ,2024)
三、TALEN技术
TALEN(Transcription ActivatorLike Effector Nuclease)是由具有DNA结合特性的TALE蛋白和FokI内切核酸酶组合成的人工工程化蛋白。TALE是在植物病原细菌Xanthomonas中发现的一类称为转录激活因子样效应子(TALE)的DNA结合蛋白,这些TALE可以调节宿主对应靶基因的转录[10]。
TALE的中心区域由34个氨基酸重复序列构成,其中32个呈现恒定状态,2个具有可变性,被称作重复可变双残基(Repeat Variable Di-residue,RVD)。RVD决定了该重复单元对特定DNA碱基的识别效能,一个RVD靶向一个核苷酸(NI - A(腺嘌呤)、HD - C(胞嘧啶)、NG - T(胸腺嘧啶)、NN - G(鸟嘌呤))[11][12]。研究人员可通过组合不同的RVD序列,依据“一位一碱基”的准则,设计出能够特异性结合任意DNA序列的TALE蛋白,进而达成精准的基因编辑。
由于FokI需进行二聚化,才能在两条链上同时造成磷酸二酯键断裂,TALEN则需要两个TALE蛋白,分别从左、右两侧结合于目标DNA,在间隔区使两个FokI结构域聚集在一起,从而诱导双链断裂(DSB)。随后,细胞会通过非同源末端连接(NHEJ)或同源重组(HDR)进行修复,进而实现基因敲除、敲入或碱基替换。
TALE技术的使用限制与优势:①TALE的重复序列致使其难以通过聚合酶链式反应(PCR)进行构建;②TALEN无法靶向甲基化的DNA,因为胞嘧啶的甲基化可能会潜在地消除TALE的结合并改变其正常RVD的识别[13];③与MN和ZFN相比,TALEN展现出高效率、低脱靶效应的特点。尽管如此,仍存在一些限制因素阻碍了TALE技术的更广泛应用。
![]()
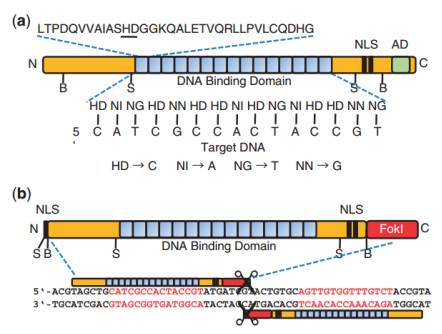
图3:TAL效应子和TALEN结构(Cermak,Tomas et al.Nucleic acids research.2011)
四、CRISPR-Cas9
2020年10月,诺贝尔化学奖授予埃马纽埃尔·沙尔庞捷(Emmanuelle Charpentier)和珍妮弗·道德纳(Jennifer A. Doudna),以表彰其发现了一种具有前景的新型基因组编辑工具——CRISPR - Cas9基因剪刀[14]。CRISPR-Cas9系统作为一种复杂的基因编辑工具,彻底革新了基因组工程领域,且为运用新型治疗策略治疗人类疾病提供了巨大潜力。
依据CRISPR-cas基因座的现行分类,CRISPR系统已被划分为六种不同类型(I - VI),每种类型均运用一组独特的Cas蛋白以及相应的crRNA形式来实现CRISPR编辑功能。不同类型中的Cas蛋白,部分分子量较大,还有部分是以复合物形式存在。其中,Ⅱ型CRISPR - Cas9系统借助单个DNA内切核酸酶Cas9识别双链DNA(dsDNA)底物,并利用不同的核酸酶结构域(HNH或RuvC)切割每条链[15]。鉴于其操作简便,该系统成为生物研究与转化应用领域中使用最为广泛的工具。
CRISPR-Cas9(Clustered Regularly Interspaced Short Palindromic Repeats CRISPR associated protein 9)最初是细菌和古菌用来抵御噬菌体入侵的适应性免疫机制,后来被改造为一种高效、低成本的基因组编辑工具。它的核心由两大部分组成:
CRISPR序列:CRISPR(成簇的规律间隔短回文重复序列,Clustered Regularly Interspaced Short Palindromic Repeats)由直接重复序列(direct repeat,DR)与间隔(spacer)序列交替排列构成,其记录了细菌过往遭遇的外源DNA片段。当细菌初次遭受病毒(如噬菌体)入侵时,CRISPR系统会捕获病毒基因的一小段序列(即原间隔序列),并将其整合至CRISPR序列的间隔区,进而形成“免疫记忆”。当相同病毒再次入侵时,CRISPR序列转录生成的CRISPR RNA(crRNA)包含间隔子(即与来自外源遗传元件的序列互补的RNA短片段),crRNA会与Cas蛋白结合,引导Cas蛋白精准识别并切割病毒DNA,以此阻止病毒的复制与感染[16]。在这一过程中,有一个需特别关注的序列,即PAM序列。在大多数CRISPR-Cas系统中,存在一个位于入侵DNA上crRNA靶序列附近、长度为2 - 5 bp的保守序列基序(称为PAM,Protospacer Adjacent Motif)。Cas蛋白在搜寻目标位点时,首先会扫描基因组中的PAM序列,只有当出现符合特定PAM模式的位点时,Cas蛋白才会进一步解旋DNA,并检查sgRNA(或crRNA)导向序列是否与之完全配对[17]。CRISPR序列中的间隔序列存储了细菌曾接触过的病毒或其他外源DNA片段,相当于细菌的“免疫档案”,反映了细菌与病毒在长期进化过程中的博弈历史。
Cas9蛋白:一种具有核酸酶活性的单体效应蛋白,其与引导RNA(gRNA)结合后,能够在特定DNA位点产生双链断裂(DSB)。Cas9由两大结构域组成:α-螺旋叶(REC)与核酸酶叶(NUC),形成双叶体(bilobed)结构。NUC叶内部包含RuvC与HNH两个依赖金属离子的核酸酶活性位点,分别负责切割DNA的非靶链和靶链[18]。
在过去的十余年里,CRISPR - Cas9系统经修饰与调适,成为真核生物基因组编辑的通用工具。该系统的工作流程大致分为以下几个阶段:
1. Cas9 - sgRNA复合体的组装:Cas9蛋白首先经由识别区域与sgRNA结合,形成稳定的Cas9-sgRNA核糖核蛋白(RNP)。结合后,Cas9发生显著的构象改变,REC叶向sgRNA方向闭合,形成一个预先装配好的“导向平台”[19]。
2. 搜索和识别目标DNA及PAM序列:Cas9-sgRNA复合体于基因组中进行搜索时,首先识别前间区序列邻近基序(PAM),例如酿脓链球菌Cas9(SpCas9)所需的5′-NGG序列。当识别到合适的PAM靶位点时,Cas9诱导目标DNA双链局部解旋,使20 nt向导序列能够与DNA的互补链形成R环(RNA - DNA异链杂交)。单链向导RNA(sgRNA)的种子区与靶DNA之间的完美互补性,对于Cas9介导的DNA靶向与切割而言是必要的[20]。
3. 双链断裂(DSB):R环形成之后,Cas9的两个核酸酶活性被激活,HNH结构域切割与sgRNA配对的DNA链(即“靶链”),RuvC结构域切割相对的非靶链[14][21][22][23]。二者协同作用产生双链断裂(DSB),通常在PAM下游3 bp处完成。
4. DSB修复:细胞随后通过非同源末端连接(NHEJ)或同源重组修复(HDR)机制进行修复,实现基因敲除、敲入或碱基替换[9]。
CRISPR/Cas9仅需sgRNA来引导DNA产生双链断裂(DSB),其结构简洁、设计便捷,同时在规模化和高通量方面具备明显优势。
CRISPR - Cas9技术存在若干局限性:①存在脱靶效应;②PAM序列的要求对编辑范围形成了限制;③需要更为有效的CRISPR/Cas9系统体内递送方法;④存在免疫毒性、DNA损伤毒性等问题。随着该技术的持续改进,这些局限性逐渐减少。例如,通过改变Cas9构象等变体[24][25][26][27]以及工程化sgRNA[28][29][30]可提升特异性和靶向切割效率。此外,还开发出了需要不同PAM序列的Cas9变体[31][32][33][34],这有助于缓解PAM序列的限制,显著增强了基因组可及性。
研究者不断探索发掘具有潜在生物技术应用价值的新型CRISPR系统。随着更多Cas9直系同源物被鉴定和表征,Cas9介导的基因组工程资源库将得到显著扩充。近年来,对CRISPR - Cas9结构和机制的研究持续深入,增进了对CRISPR - Cas9作用机制的理解,为基于结构的理性设计构建了框架。与此同时,借助生物信息学工具,人们努力提高CRISPR - Cas9的效率,并尽可能降低其在人类健康和治疗领域的脱靶效应。2023年11月,全球首款基于CRISPR/Cas9技术的基因编辑药物Casgevy在英国获批上市,这是由CRISPR Therapeutics和Vertex联合推出的一款自体、ex vivo CRISPR/Cas9基因编辑药物,用于治疗12岁及以上输血依赖性β地中海贫血(TDT)或伴有复发性血管闭塞危象(VOC)的镰状细胞病(SCD)患者。
CRISPR介导的基因靶向技术,凭借其易于编程和广泛适用的特性,已被研究人员用于开展一系列拓展应用。扩展编辑功能主要涵盖以下几个方向:
(一)基因激活(CRISPRa)和抑制(CRISPRi):研究者将dCas9(核酸酶失活突变形式)与转录激活子或阻遏子融合,借助sgRNA的特异性,将转录激活因子或阻遏子引导并定位到特定的DNA序列,以抑制(CRISPRi)或激活(CRISPRa)靶基因的转录[35][36],进而实现对基因表达的精确控制。这种方法推动了基因功能和调控网络的研究。常用的转录激活因子包括VP64、VPR等,常用的转录抑制因子有KRAB、SALL1 - SDS3等。
(二)碱基编辑器((base editor, BE):这类编辑器包含两个主要组件:与脱氨酶融合的Cas9n以及能与特定DNA序列结合的sgRNA[37]。其中,Cas9n仅保留一个活性核酸酶域和与sgRNA结合的能力。该融合体可利用sgRNA的编程能力,但不会诱导双链DNA断裂,而是通过脱氨酶介导碱基转换。目前已创建了两种类型的碱基编辑器。①胞苷碱基编辑器(CBE):由胞苷脱氨酶和Cas9n组成,利用sgRNA靶向特定的基因组序列,将单链DNA序列中一定窗口范围内的脱氧胞苷(C)酶促脱氨为脱氧尿苷(U)[38]。②腺苷碱基编辑器(ABE):由腺苷脱氨酶和Cas9n组成,介导基因组DNA中A·T向G·C的转化[39]。对于大多数已知的遗传疾病,校正靶基因座中的点突变时,碱基编辑器会是更为有效的策略。
![]()
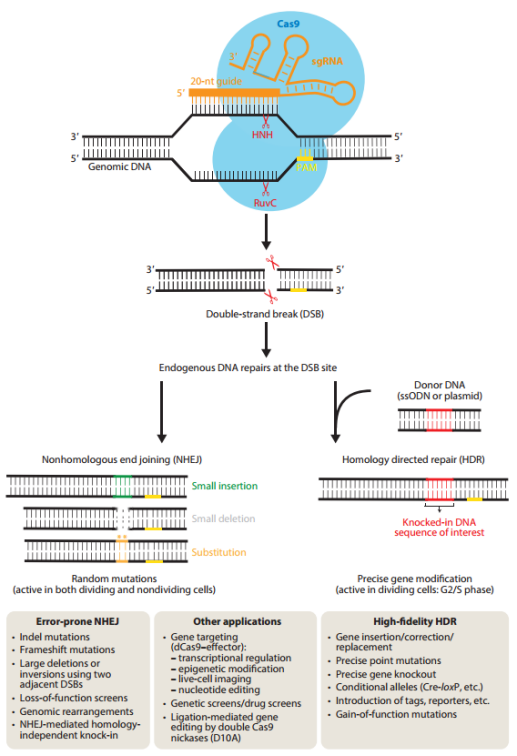
图4:CRISPR-Cas9介导的基因组编辑的机制(Jiang F, Doudna JA . Annu. Rev. Biophys. 2017)
五、CRISPR-Cas13系统
CRISPR - Cas13系统作为一种RNA碱基编辑工具,具备重要的研究价值。Cas13归属于CRISPR - Class 2 type VI系统,该type VI系统的效应蛋白在细菌与古菌中主要发挥抵御RNA病毒或转座子RNA的功能,其作用机制专门针对单链RNA而非DNA[40][41],这为新型RNA靶向工具的开发提供了可能性。Cas13蛋白的核心催化域为HEPN(Higher - Eukaryotes and Prokaryotes Nucleotide - binding)RNase双结构域。HEPN结构域含有构成金属非依赖性内切RNA酶活性位点的保守基序,其与任何已知的DNA核酸酶结构域缺乏同源性[42]。
Cas13的crRNA仅包含一个茎环(结构域)和22 - 30 nt的间隔序列,该间隔序列与单链RNA完全配对后才能触发HEPN催化活性。目标RNA与crRNA配对后,Cas13进入激活构象,HEPN双域相互靠近,产生RNA切割活性[43]。
CRISPR/Cas 13因其特异性靶向和降解病毒RNA的能力,成为一种颇具前景的抗病毒工具。与传统的抗病毒策略不同,Cas 13在RNA水平发挥作用,提供了一种广谱且可编程的方法来对抗RNA病毒,例如抗SARS-CoV-2病毒的相关研究[44][45][46];抗登革热病毒的相关研究[47][48][49];抗人类免疫缺陷病毒(HIV)的相关研究[50][51]。
Cas13的RNA靶向和RNA切割行为为RNA检测和诊断应用提供了作用机制,目前已开发出HEPN结构域失活的不同Cas13版本,这些版本作为可编程的RNA结合蛋白可应用于RNA检测[52]、RNA编辑[53]、RNA调控[54]等领域。
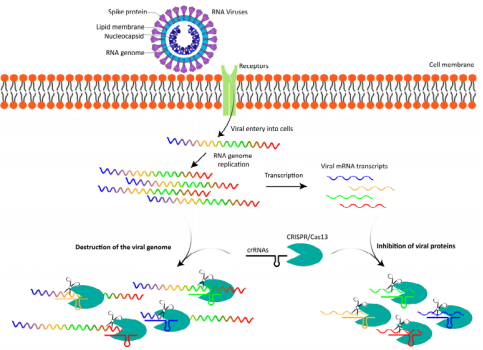
![]() 图5:Cas13介导的RNA病毒复制抑制示意图(Tan X, Li J, Cui B, et al. Genes (Basel). 2025)
图5:Cas13介导的RNA病毒复制抑制示意图(Tan X, Li J, Cui B, et al. Genes (Basel). 2025)
六、结束语
在过去十年里,基因编辑技术在优化与应用领域取得了显著进步。随着这些技术不断优化,锌指核酸酶(ZFN)、转录激活因子样效应物核酸酶(TALENs)以及规律成簇间隔短回文重复序列及其相关系统(CRISPR - Cas)已进入人体临床试验阶段,且部分基于基因编辑技术的基因治疗药物已获批上市。
截至目前,这些技术的多数临床应用主要集中于离体基因编辑疗法。离体编辑对于某些疾病,例如镰状细胞病,效果十分显著。
然而,基因编辑的理想应用是对体内细胞进行修饰,但基因编辑技术在体内的应用受到脱靶、编辑效率低下等问题的限制。目前针对这些问题所开展的研究,或许会为这些核酸酶介导技术的临床应用开拓新的途径。
七、参考文献列表
[1] Benusiglio, P. R., Fallet, V., Sanchis-Borja, M., Coulet, F., & Cadranel, J. (2021). Lung cancer is also a hereditary disease. European respiratory review : an official journal of the European Respiratory Society, 30(162), 210045.
[2] Claussnitzer, M., Cho, J. H., Collins, R., Cox, N. J., Dermitzakis, E. T., Hurles, M. E., Kathiresan, S., Kenny, E. E., Lindgren, C. M., MacArthur, D. G., North, K. N., Plon, S. E., Rehm, H. L., Risch, N., Rotimi, C. N., Shendure, J., Soranzo, N., & McCarthy, M. I. (2020). A brief history of human disease genetics. Nature, 577(7789), 179–189.
[3] Thierry,A. and Dujon,B. (1992) Nested chromosomal fragmentation in yeast using the meganuclease I-Sce I: a new method for physical mapping of eukaryotic genomes. Nucleic Acids Res., 20, 5625–5631.
[4] Epinat, J. C., Arnould, S., Chames, P., Rochaix, P., Desfontaines, D., Puzin, C., Patin, A., Zanghellini, A., Pâques, F., & Lacroix, E. (2003). A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic acids research, 31(11), 2952–2962.
[5] Mandip KC, Steer CJ (2019) A new era of gene editing for the treatment of human diseases. Swiss Med Wkly 149:w20021
[6] Walker-Daniels J (2013) CRISPR and genomic engineering. Mater Methods 3:164
[7] Sun, Z., Wang, M., Han, S., Ma, S., Zou, Z., Ding, F., et al. (2018). Production of hypoallergenic milk from DNA-free beta-lactoglobulin (BLG) gene knockout cow using zinc-finger nucleases mRNA. Sci. Rep. 8, 15430.
[8] Doyon, Y., Vo, T. D., Mendel, M. C., Greenberg, S. G., Wang, J., Xia, D. F., Miller, J. C., Urnov, F. D., Gregory, P. D., & Holmes, M. C. (2011). Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nature methods, 8(1), 74–79.
[9] Lessard S, Rimmelé P, Ling H, et al. Zinc finger nuclease-mediated gene editing in hematopoietic stem cells results in reactivation of fetal hemoglobin in sickle cell disease. Sci Rep. 2024;14(1):24298. Published 2024 Oct 16.
[10] Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng, G., Zhang, F., et al.(2012). A transcription activator-like effector toolbox for genome engineering. Nat.Protoc. 7, 171–192.
[11] Moscou,M.J. and Bogdanove,A.J. (2009) A simple cipher governs DNA recognition by TAL effectors. Science, 326, 1501.
[12] Boch,J., Scholze,H., Schornack,S., Landgraf,A., Hahn,S., Kay,S.,Lahaye,T., Nickstadt,A. and Bonas,U. (2009) Breaking the code
of DNA binding specificity of TAL-type III effectors. Science,326, 1509–1512.
[13] Deng, P., Carter, S., & Fink, K. (2019). Design, Construction, and Application of Transcription Activation-Like Effectors. Methods in molecular biology (Clifton, N.J.), 1937, 47–58.
[14] Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., & Charpentier, E. (2012). A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science (New York, N.Y.), 337(6096), 816–821.
[15] Tang, Y., and Fu, Y. (2018). Class 2 CRISPR/Cas: An expanding biotechnology toolbox for and beyond genome editing. Cell. Biosci. 8, 59. doi:10.1186/s13578-018- 0255-x
[16] Wiedenheft, B., Sternberg, S. H., & Doudna, J. A. (2012). RNA-guided genetic silencing systems in bacteria and archaea. Nature, 482(7385), 331–338.
[17] Bolotin, A., Quinquis, B., Sorokin, A., & Ehrlich, S. D. (2005). Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology (Reading, England), 151(Pt 8), 2551–2561.
[18] Jiang, F., & Doudna, J. A. (2017). CRISPR-Cas9 Structures and Mechanisms. Annual review of biophysics, 46, 505–529.
[19] Jinek, M., Jiang, F., Taylor, D. W., Sternberg, S. H., Kaya, E., Ma, E., Anders, C., Hauer, M., Zhou, K., Lin, S., Kaplan, M., Iavarone, A. T., Charpentier, E., Nogales, E., & Doudna, J. A. (2014). Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science (New York, N.Y.), 343(6176), 1247997.
[20] Wu, X., Scott, D. A., Kriz, A. J., Chiu, A. C., Hsu, P. D., Dadon, D. B., Cheng, A. W., Trevino, A. E., Konermann, S., Chen, S., Jaenisch, R., Zhang, F., & Sharp, P. A. (2014). Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nature biotechnology, 32(7), 670–676.
[21] Jiang, W., Bikard, D., Cox, D., Zhang, F., & Marraffini, L. A. (2013). RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature biotechnology, 31(3), 233–239. https://doi.org/10.1038/nbt.2508
[22] Jiang, F., Taylor, D. W., Chen, J. S., Kornfeld, J. E., Zhou, K., Thompson, A. J., Nogales, E., & Doudna, J. A. (2016). Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. Science (New York, N.Y.), 351(6275), 867–871.
[23] Palermo, G., Miao, Y., Walker, R. C., Jinek, M., & McCammon, J. A. (2016). Striking Plasticity of CRISPR-Cas9 and Key Role of Non-target DNA, as Revealed by Molecular Simulation-s. ACS central science, 2(10), 756–763.
[24] Kleinstiver, B. P., Pattanayak, V., Prew, M. S., Tsai, S. Q., Nguyen, N. T., Zheng, Z., & Joung, J. K. (2016). High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature, 529(7587), 490–495.
[25] Slaymaker, I. M., Gao, L., Zetsche, B., Scott, D. A., Yan, W. X., & Zhang, F. (2016). Rationally engineered Cas9 nucleases with improved specificity. Science (New York, N.Y.), 351(6268), 84–88.
[26] Chen, J. S., Dagdas, Y. S., Kleinstiver, B. P., Welch, M. M., Sousa, A. A., Harrington, L. B., Sternberg, S. H., Joung, J. K., Yildiz, A., & Doudna, J. A. (2017). Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature, 550(7676), 407–410.
[27] Liu, M. S., Gong, S., Yu, H. H., Jung, K., Johnson, K. A., & Taylor, D. W. (2020). Engineered CRISPR/Cas9 enzymes improve discrimination by slowing DNA cleavage to allow release of off-target DNA. Nature communications, 11(1), 3576.
[28] Doench, J. G., Hartenian, E., Graham, D. B., Tothova, Z., Hegde, M., Smith, I., Sullender, M., Ebert, B. L., Xavier, R. J., & Root, D. E. (2014). Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature biotechnology, 32(12), 1262–1267.
[29] Kocak, D. D., Josephs, E. A., Bhandarkar, V., Adkar, S. S., Kwon, J. B., & Gersbach, C. A. (2019). Increasing the specificity of CRISPR systems with engineered RNA secondary structures. Nature biotechnology, 37(6), 657–666.
[30] Nelson, J. W., Randolph, P. B., Shen, S. P., Everette, K. A., Chen, P. J., Anzalone, A. V., An, M., Newby, G. A., Chen, J. C., Hsu, A., & Liu, D. R. (2022). Engineered pegRNAs improve prime editing efficiency. Nature biotechnology, 40(3), 402–410.
[31] Kleinstiver, B. P., Prew, M. S., Tsai, S. Q., Topkar, V. V., Nguyen, N. T., Zheng, Z., Gonzales, A. P., Li, Z., Peterson, R. T., Yeh, J. R., Aryee, M. J., & Joung, J. K. (2015). Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 523(7561), 481–485.
[32] Nishimasu, H., Shi, X., Ishiguro, S., Gao, L., Hirano, S., Okazaki, S., Noda, T., Abudayyeh, O. O., Gootenberg, J. S., Mori, H., Oura, S., Holmes, B., Tanaka, M., Seki, M., Hirano, H., Aburatani, H., Ishitani, R., Ikawa, M., Yachie, N., Zhang, F., … Nureki, O. (2018). Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science (New York, N.Y.), 361(6408), 1259–1262.
[33] Hu, J. H., Miller, S. M., Geurts, M. H., Tang, W., Chen, L., Sun, N., Zeina, C. M., Gao, X., Rees, H. A., Lin, Z., & Liu, D. R. (2018). Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 556(7699), 57–63.
[34] Walton, R. T., Christie, K. A., Whittaker, M. N., & Kleinstiver, B. P. (2020). Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science (New York, N.Y.), 368(6488), 290–296.
[35] Gilbert, L. A., Larson, M. H., Morsut, L., Liu, Z., Brar, G. A., Torres, S. E., Stern-Ginossar, N., Brandman, O., Whitehead, E. H., Doudna, J. A., Lim, W. A., Weissman, J. S., & Qi, L. S. (2013). CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 154(2), 442–451.
[36] Sander, J. D., & Joung, J. K. (2014). CRISPR-Cas systems for editing, regulating and targeting genomes. Nature biotechnology, 32(4), 347–355.
[37] Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., & Liu, D. R. (2016). Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 533(7603), 420–424.
[38] Gilbert, L. A., Horlbeck, M. A., Adamson, B., Villalta, J. E., Chen, Y., Whitehead, E. H., Guimaraes, C., Panning, B., Ploegh, H. L., Bassik, M. C., Qi, L. S., Kampmann, M., & Weissman, J. S. (2014). Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell, 159(3), 647–661.
[39] Gaudelli, N. M., Komor, A. C., Rees, H. A., Packer, M. S., Badran, A. H., Bryson, D. I., & Liu, D. R. (2017). Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 551(7681), 464–471.
[40] Abudayyeh, O. O., Gootenberg, J. S., Konermann, S., Joung, J., Slaymaker, I. M., Cox, D. B., Shmakov, S., Makarova, K. S., Semenova, E., Minakhin, L., Severinov, K., Regev, A., Lander, E. S., Koonin, E. V., & Zhang, F. (2016). C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science (New York, N.Y.), 353(6299), aaf5573.
[41] Shmakov, S., Abudayyeh, O. O., Makarova, K. S., Wolf, Y. I., Gootenberg, J. S., Semenova, E., Minakhin, L., Joung, J., Konermann, S., Severinov, K., Zhang, F., & Koonin, E. V. (2015). Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems. Molecular cell, 60(3), 385–397.
[42] Anantharaman, V., Makarova, K. S., Burroughs, A. M., Koonin, E. V., & Aravind, L. (2013). Comprehensive analysis of the HEPN superfamily: identification of novel roles in intra-genomic conflicts, defense, pathogenesis and RNA processing. Biology direct, 8, 15. https://doi.org/10.1186/1745-6150-8-15
[43] Tambe, A., East-Seletsky, A., Knott, G. J., Doudna, J. A., & O'Connell, M. R. (2018). RNA Binding and HEPN-Nuclease Activation Are Decoupled in CRISPR-Cas13a. Cell reports, 24(4), 1025–1036.
[44] Abbott, T. R., Dhamdhere, G., Liu, Y., Lin, X., Goudy, L., Zeng, L., Chemparathy, A., Chmura, S., Heaton, N. S., Debs, R., Pande, T., Endy, D., La Russa, M. F., Lewis, D. B., & Qi, L. S. (2020). Development of CRISPR as an Antiviral Strategy to Combat SARS-CoV-2 and Influenza. Cell, 181(4), 865–876.e12.
[45] Zeng, L., Liu, Y., Wrynla, X. H., Abbott, T. R., Han, M., Zhu, Y., Chemparathy, A., Lin, X., Chen, X., Wang, H., Rane, D. A., Spatz, J. M., Jain, S., Rustagi, A., Pinsky, B., Zepeda, A. E., Kadina, A. P., Walker, J. A., 3rd, Holden, K., Temperton, N., Qi, L. S. (2022). Broad-spectrum CRISPR-mediated inhibition of SARS-CoV-2 variants and endemic coronaviruses in vitro. Nature communications, 13(1), 2766.
[46] Hussein, M., Andrade Dos Ramos, Z., Vink, M. A., Kroon, P., Yu, Z., Enjuanes, L., Zuñiga, S., Berkhout, B., & Herrera-Carrillo, E. (2023). Efficient CRISPR-Cas13d-Based Antiviral Strategy to Combat SARS-CoV-2. Viruses, 15(3), 686.
[47] Li, H., Wang, S., Dong, X., Li, Q., Li, M., Li, J., Guo, Y., Jin, X., Zhou, Y., Song, H., & Kou, Z. (2020). CRISPR-Cas13a Cleavage of Dengue Virus NS3 Gene Efficiently Inhibits Viral Replication. Molecular therapy. Nucleic acids, 19, 1460–1469.
[48] Singsuksawat, E., Onnome, S., Posiri, P., Suphatrakul, A., Srisuk, N., Nantachokchawapan, R., Praneechit, H., Sae-Kow, C., Chidpratum, P., Sa-Ngiamsuntorn, K., Hongeng, S., Avirutnan, P., Duangchinda, T., & Siridechadilok, B. (2021). Potent programmable antiviral against dengue virus in primary human cells by Cas13b RNP with short spacer and delivery by VLP. Molecular therapy. Methods & clinical development, 21, 729–740.
[49] Basu, M., Zurla, C., Auroni, T. T., Vanover, D., Chaves, L. C. S., Sadhwani, H., Pathak, H., Basu, R., Beyersdorf, J. P., Amuda, O. O., Elsharkawy, A., Mosur, V., Arthur, R. A., Claussen, H., Sasser, L. E., Wroe, J. A., Peck, H. E., Kumar, M., Brinton, M. A., & Santangelo, P. J. (2024). mRNA-encoded Cas13 can be used to treat dengue infections in mice. Nature microbiology, 9(8), 2160–2172.
[50] Yin, L., Zhao, F., Sun, H., Wang, Z., Huang, Y., Zhu, W., Xu, F., Mei, S., Liu, X., Zhang, D., Wei, L., Cen, S., Hu, S., Liang, C., & Guo, F. (2020). CRISPR-Cas13a Inhibits HIV-1 Infection. Molecular therapy. Nucleic acids, 21, 147–155.
[51] Nguyen, H., Wilson, H., Jayakumar, S., Kulkarni, V., & Kulkarni, S. (2021). Efficient Inhibition of HIV Using CRISPR/Cas13d Nuclease System. Viruses, 13(9), 1850.
[52] Abudayyeh, O. O., Gootenberg, J. S., Essletzbichler, P., Han, S., Joung, J., Belanto, J. J., Verdine, V., Cox, D. B. T., Kellner, M. J., Regev, A., Lander, E. S., Voytas, D. F., Ting, A. Y., & Zhang, F. (2017). RNA targeting with CRISPR-Cas13. Nature, 550(7675), 280–284.
[53] Cox, D. B. T., Gootenberg, J. S., Abudayyeh, O. O., Franklin, B., Kellner, M. J., Joung, J., & Zhang, F. (2017). RNA editing with CRISPR-Cas13. Science (New York, N.Y.), 358(6366), 1019–1027.
[54] Konermann, S., Lotfy, P., Brideau, N. J., Oki, J., Shokhirev, M. N., & Hsu, P. D. (2018). Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell, 173(3), 665–676.e14.